こちらはStanアドベントカレンダー17日目の記事です。
この記事はHainesの記事を参考に作成してます。より詳細な内容を知りたい人はこっち
ここでは社会・行動科学にありがちな主観指標と行動指標の相関を、生成モデルの観点から推定することを目指す。このアプローチには、
①前提を明示的にできる
②Priorの設定をドメイン知識に合わせれる
③不確実性を定量化できる
といった利点がある。
2値で回答可能な質問紙Aをとるとする。そんで反応時間のような行動指標Bもとるとする。
ここで知りたいのはこの質問紙Aと行動指標Bの相関であるとする。
よくあるのは、質問紙Aのアイテムを合計して計算する平均値と行動指標Bの平均値とかを使って相関を出す方法だ。直観的にもわかると思うが、このありがちなアプローチには「これらの平均値は完全な精度で推定できている」という強めの仮定を置いていることを意味する。
そこで、測定誤差を含める形でうまいこと推定するStanコードをここでは紹介する。
モデル内のパラメータ関係+観測データとモデルパラメータの関係を記述可能なデータ生成プロセスを特定することが目的となる。
質問紙A
質問紙についてはベルヌーイ試行として各アイテム\(t\)に対する反応にベルヌーイ分布を適用する。ベルヌーイモデルは以下の通りシンプルなものだ。
\[ Pr(x_{i,t} = 1) = p_i = 1 - Pr(x_{i,t} = 0) = 1- q_i \]
\(p_i\)=個人\(i\)の成功確率、\(q_i\)=個人\(i\)の失敗確率である。簡単!
ここでは\(p_i\)を以下のように定義する。
\[ p_i = \cfrac{1}{1+exp(-\theta_i)} \]
\(\theta_i\)は個人\(i\)の回答確率と対応する能力と解釈でき(-∞~∞)、\(\theta_i\)にロジスティック関数かました\(p_i\)は0~1の値をとる。
知りたいのは、観測された確率\(p_i\)の背後に存在する\(\theta_i\)である。
ここからは個人レベルの生成プロセスについて見ていく。これは以下のように階層的なパラメータを設定すればよい。階層モデルの説明はStanアドカレ9日目の資料なりなんなり、たくさんあるのでそれを見ること。
\[
\theta_i ~ Normal(\mu_{\theta}, \sigma_{\theta})
\]
さらに集団平均と標準偏差パラメータに関する生成プロセス(\(theta_i\)のハイパーパラメータということもある)を設定する必要があり、ここでは
\[
\mu_{\theta} ~ Normal(0, 1)
\]
\[ log(\sigma_{\theta}) ~ Normal(0, 1) \]
とする。
まとめると
\[ Y_i = \cfrac{1}{1+exp(-\theta_i)} \]
\[ \theta_i ~ Normal(\mu_{\theta}, \sigma_{\theta}) \] \[ \mu_{\theta} ~ normal(0,1) \] \[ log(\sigma_{\theta}) ~ normal(0,1) \]
以上が質問紙に対する反応の生成プロセスを記述したものだ。次は行動課題、ここでは反応時間課題のものを考えてみよう。
反応時間!!!
各個人\(i\)の試行\(t\)における反応時間は以下の正規分布に従う
\[ RT_{i,t} ~ Normal(\delta_i, \sigma_i) \]
質問紙(での\(\theta_i\))と同様に各パラメータに事前分布を階層的に置くと
\[ \delta_i ~ Normal(\mu_{\delta}, \sigma_{\delta}) \] \[ log(\sigma_i) ~ Normal(\mu_{\sigma}, \sigma_{\sigma}) \]
さらに各生成プロセスを設定する必要がある。ここでは以下のようにする。
\[ \mu_{\delta} ~ Normal(1, 1) \] \[ log(\sigma_{\delta}) ~ Normal(0, 0.5) \] \[ \mu_{\sigma} ~ Normal(-1, 0.2) \] \[ log(\sigma_{\sigma}) ~ Normal(0,0.5) \]
反応時間は正規分布に従わないし、非負だし、200ms以上が典型的。だが、ここではそこらへんの要素は単純化のため省く。
標準偏差の仮定に半コーシー分布を置いたり、ドメイン知識をいかした拡張をするのも興味深いだろう。
とりあえず行動指標についての生成プロセスをまとめると
\[ RT_{i,t} ~ Normal(\delta_i, \sigma_i) \]
\[ \delta_i ~ Normal(\mu_{\delta}, \sigma_{\delta}) \] \[ \mu_{\delta} ~ Normal(1, 1) \] \[ log(\sigma_{\delta}) ~ Normal(0, 0.5) \] \[ log(\sigma_i) ~ Normal(\mu_{\sigma}, \sigma_{\sigma}) \] \[ \mu_{\sigma} ~ Normal(-1, 0.2) \] \[ log(\sigma_\sigma) ~ Normal(0,0.5) \]
このようになる。ながいな。コードはもっとながいぞ、各位覚悟するべし。
質問紙と反応時間データの混合生成モデル
知りたいのは観測されたアイテムの平均値×反応時間の平均値、ではなくその背後にある\(\theta_i\)と\(\delta_i\)の相関である。
二つのパラメータが多変量正規分布から生成されたという仮定を置く
\[ \theta_i ~ Normal(\mu_{\theta}, \sigma_{\theta}) \] \[ \delta_i ~ Normal(\mu_{\delta}, \sigma_{\delta}) \]
から
\[ \begin{bmatrix} \theta_i \\ \delta_i \end{bmatrix} ~ MVNormal(\begin{bmatrix} \mu_{\theta} \\ \mu_{\delta} \end{bmatrix}, S) \]
とする。
ここでのSは、集団レベルの標準偏差と2×2の相関行列Rに分解可能な共分散行列である。
\[ S = \begin{pmatrix} \sigma_{\theta} && 0 \\ 0 && \sigma_{\delta} \end{pmatrix} * \begin{pmatrix} 1 && \rho_{\theta,\delta} \\ \rho_{\theta,\delta} && 1 \end{pmatrix} * \begin{pmatrix} \sigma_{\theta} && 0 \\ 0 && \sigma_{\delta} \end{pmatrix} \]
ここでの相関行列にはLKJcorr(1)の無情報事前分布を設定しよう。
以上をまとめると以下のようなStanコードになる
data {
int N; // 参加者
int N_items; // アイテム数
int T; // 反応時間課題の試行数
real RT[N, T]; // 各参加者、各試行での反応時間
int Y[N, N_items]; // 各参加者、各アイテムでの反応
}
parameters {
// 集団レベルのパラメータ
// 計算の効率化のためにコレスキー分解を利用
// 下三角行列のみの相関行列を作成
// 詳しくはManual:https://mc-stan.org/docs/2_21/functions-reference/cholesky-lkj-correlation-distribution.html
cholesky_factor_corr[2] R_chol;
// SDs
vector<lower=0>[2] pars_sigma; // non-centered parameterization用=l + s*a の形に書き下す方法
real<lower=0> sigma_SD; // SDのsd
// 平均
vector[2] pars_mu; // [1] = 質問紙、[2] = 反応時間
real sigma_mu; // 反応時間の標準偏差の平均
// 個人レベルのパラメータ
matrix[2,N] pars_pr; // [1,i]=質問紙の個人差、[2,i]=反応時間の個人差
vector[N] sigma_pr; // 反応時間の標準偏差に関するnon-centered用
}
transformed parameters {
// non-centered parameterization専用
matrix[2,N] pars_tilde;
// 個人レベルのパラメータ
vector[N] theta;
vector[N] delta;
vector[N] sigma;
// non-centered parameterization=l + s*a の形に書き下す方法
// diag_pre_multiply()
// =pars_sigmaを対称行列にしてR_cholとの積を計算-> s*aの形を提供
// 例:beta ~ normal(mu_beta, sigma_beta)
// → beta = mu_beta + sigma_beta*beta_raw; ... model{beta_raw ~ normal(0,1)}
// https://mc-stan.org/docs/2_18/stan-users-guide/reparameterization-section.html
pars_tilde = diag_pre_multiply(pars_sigma, R_chol) * pars_pr;
// Compute individual-level parameters from non-centered parameterization
for (i in 1:N) {
theta[i] = pars_mu[1] + pars_tilde[1, i];
delta[i] = pars_mu[2] + pars_tilde[2, i];
sigma[i] = exp(sigma_mu + sigma_SD * sigma_pr[i]);
}
}
model {
// コレスキー用相関行列に無情報事前分布を設定
R_chol ~ lkj_corr_cholesky(1);
// 集団レベルの平均についての事前分布
pars_mu[1] ~ normal(0, 1); // 質問紙
pars_mu[2] ~ normal(1, 1); // 反応時間
sigma_mu ~ normal(-1, .2); // 反応時間の標準偏差
// 集団レベルの標準偏差についての事前分布
pars_sigma[1] ~ normal(0, 1); // 質問紙
pars_sigma[2] ~ normal(0, .5); // 反応時間
sigma_SD ~ normal(0, .5); // 反応時間の標準偏差
// 個人レベルのパラメータの事前分布
to_vector(pars_pr) ~ normal(0, 1); // [1,i]=質問紙の個人差、[2,i]=反応時間の個人差
to_vector(sigma_pr) ~ normal(0, 1); // 反応時間の標準偏差の個人差
// For each subject
for (i in 1:N) {
// 反応時間のモデル
RT[i,1:T] ~ normal(delta[i], sigma[i]);
// 質問紙のモデル
Y[i,:] ~ bernoulli_logit(theta[i]);
}
}
generated quantities {
corr_matrix[2] R;
// L*L' で正定値対称相関行列が作れる
R = R_chol * R_chol';
} 上記のStanコードは相関行列の計算にコレスキー分解を用いること、階層モデルに使われがちなNon-centered parameterizationが活用されていることによって計算が効率化されているのだが、基本的にはこの記事で記述してきたモデルと等価のものとなっている。安心してほしい。もっと読みやすい等価のはずのStanコードはうまくいかなかったのでボツとした。すまない。
それでは回してみよう。
以下は100人の参加者に、10のアイテム、50試行の反応時間課題でのシミュレーションデータを0-1までを20回ずつで刻んだ相関係数で試す。
library(mvtnorm)
library(doParallel)
library(rstan)
library(hBayesDM)
set.seed(2521)
# 参加者・アイテム・試行数
n_subj <- 100
n_items <- 10
n_trials <- 50
# 0ー1の相関を20刻みで
true_r <- seq(0,1,length.out = 20)
# Stan コード読み込み
m_joint_RT_Bern <- stan_model("m_joint_R.stan")
# 並列化
cl <- makeCluster(4)
registerDoParallel(cl)
# 相関ごとの計算
joint_dat <- foreach(r=seq_along(true_r), .combine = "rbind",
.packages = c("mvtnorm", "dplyr",
"foreach", "rstan", "hBayesDM")) %dopar% {
# 相関・共分散行列の作成
M <- c(0, .8) # theta + deltaの集団レベル平均
SD <- c(1, .1) # 〃の集団レベル標準偏差
R <- matrix(c(1, true_r[r], true_r[r], 1), nrow = 2)
S <- diag(SD) %*% R %*% diag(SD)
# 多変量正規分布から個人レベル記述
pars <- rmvnorm(n_subj, M, S) %>%
as.data.frame()
theta <- pars[,1] # for ベルヌ―イ
delta <- pars[,2] # for 正規分布
sigma <- rnorm(n_subj, .4, .05) # for 正規分布
# ベルヌーイ生成モデル
X_Q_all <- foreach(i=1:n_subj, .combine = "rbind") %do% {
p <- 1/(1 + exp(-theta[i])) # ロジスティック変換
rbinom(n_items, 1, prob = p)
}
# 反応時間モデル(正規分布
X_RT_all <- foreach(i=1:n_subj, .combine = "rbind") %do% {
rnorm(n_trials, delta[i], sigma[i])
}
# 観測得点の集団平均
X_bar_Q <- mean(rowMeans(X_Q_all))
X_bar_RT <- mean(rowMeans(X_RT_all))
# 個人レベルの観測得点
X_Q <- rowMeans(X_Q_all)
X_RT <- rowMeans(X_RT_all)
# 観測された相関+50%CI
obs_cor <- cor.test(X_Q, X_RT, conf.level = .5)
obs_r <- obs_cor$estimate
obs_ci <- obs_cor$conf.int
# Stan data
stan_data <- list(N = n_subj,
N_items = n_items,
T = n_trials,
Y = X_Q_all,
RT = X_RT_all)
# Fit
fit_joint <- sampling(m_joint_RT_Bern,
data = stan_data,
iter = 1000,
warmup = 200,
chains = 1,
cores = 1,
seed = 2521)
pars <- rstan::extract(fit_joint) # パラメータ抽出
# 相関+50% HDI
bayes_r <- mean(pars$R[,1,2])
hdi <- hBayesDM::HDIofMCMC(pars$R[,1,2], credMass = .5)
# まとめる
data.frame(true_r = true_r[r],
obs_r = obs_r,
obs_lo = obs_ci[1],
obs_hi = obs_ci[2],
bayes_r = bayes_r,
bayes_lo = hdi[1],
bayes_hi = hdi[2])
}
# 並列やめ
stopCluster(cl)
# 図示
qplot() +
geom_line(aes(x = true_r, y = true_r), col = I("black"),
linetype = 2, size = 1) +
geom_ribbon(aes(x = true_r,
ymin = joint_dat$obs_lo,
ymax = joint_dat$obs_hi,
fill = I("gray")), alpha = .2) +
geom_ribbon(aes(x = true_r,
ymin = joint_dat$bayes_lo,
ymax = joint_dat$bayes_hi,
fill = I("#8F2727")), alpha = .2) +
geom_line(aes(x = true_r, y = joint_dat$obs_r, col = I("gray"))) +
geom_line(aes(x = true_r, y = joint_dat$bayes_r, col = I("#8F2727"))) +
annotate("text", x = .8, y = .45, label = "Observed",
color = "gray", size = 5) +
annotate("text", x = .85, y = .75, label = "Generative",
color = "#8F2727", size = 5) +
coord_cartesian(xlim = c(0,1), ylim = c(0,1)) +
ggtitle("主観報告×行動課題") +
xlab("真の相関係数") +
ylab("推定された相関") +
theme_minimal(base_size = 15) +
theme(panel.grid = element_blank())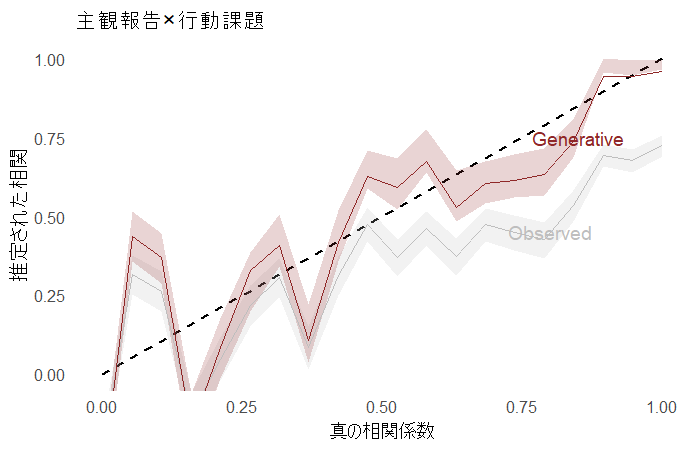
生成モデルの結果を見ると推定値とシミュレーションの値が、シンプルな相関係数よりも近いものとなっている。やったね。
ここで紹介されてるコードはいろいろと改造がききやすそうなので、自分の知りたい現象にフィットする確率分布をあてはめたり、事前分布の設定をいろいろと試したりして同様の検証を行うとよい。
Enjoy Stan!
